※本ページの内容はテキストの範囲外となります
本章では勉強会であった質問事項などを整理しています。
- Rcmdrのメニューから[データ]-[データセットのロード…]を実行する
- ファイルダイアログで「
外来患者ストレス.RData」ファイルを選択する - アクティブデータセットが
PatientStressになっていることを確認する
箱ひげ図のサマリを知る方法
R Commanderの箱ひげ図(Boxplot()関数)は外れ値のインデックスを出力することは可能ですが、四分位数などの箱ひげ図に関するサマリを出力できません。
グラフ - 箱ひげ図
car::Boxplot(ストレス反応得点 ~ 性別, data = PatientStress)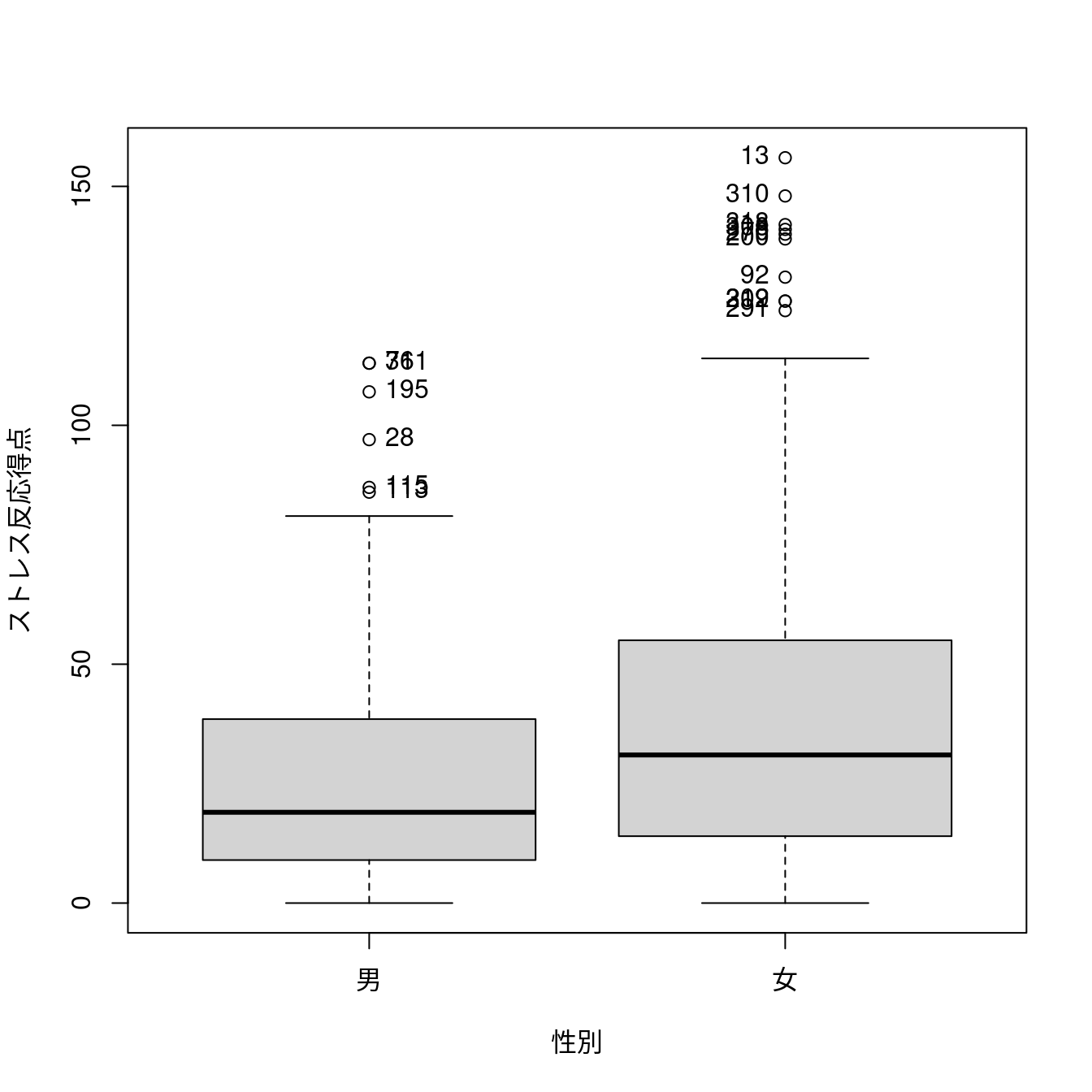
[1] "28" "76" "113" "115" "195" "311" "13" "92" "200" "209" "270" "291"
[13] "308" "310" "312" "318"そこで、サマリを関数の返り値として出力できるRの標準的箱ひげ図関数boxplot()関数を用います。ただし、サマリは非表示の返り値になっているためprint()関数を用いて明示的に出力する必要があります。
Boxplot()関数とboxplot()関数の関係
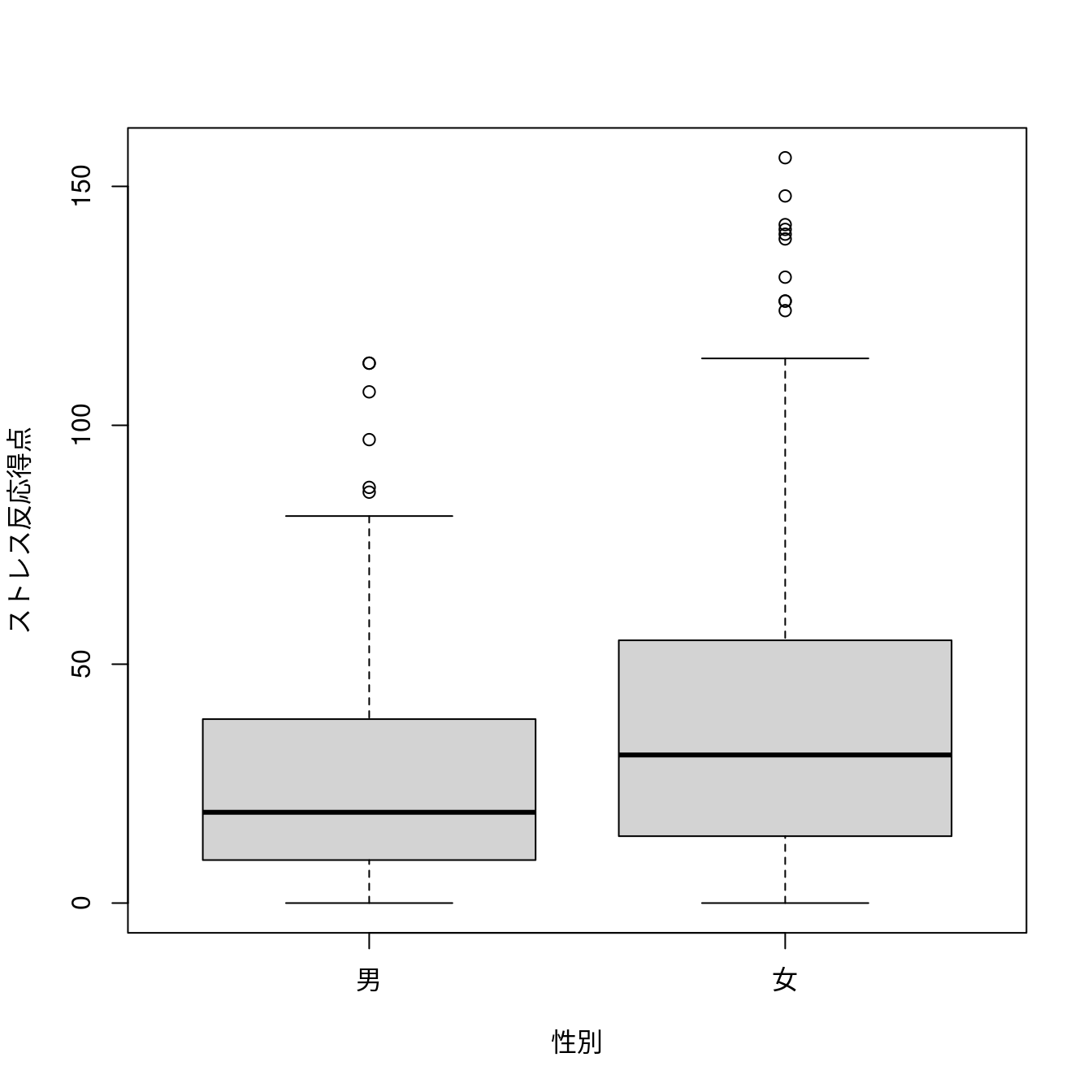
$stats
[,1] [,2]
[1,] 0.0 0
[2,] 9.0 14
[3,] 19.0 31
[4,] 38.5 55
[5,] 81.0 114
$n
[1] 119 218
$conf
[,1] [,2]
[1,] 14.72727 26.61254
[2,] 23.27273 35.38746
$out
[1] 97 113 86 87 107 113 156 131 139 126 140 124 141 148 126 142
$group
[1] 1 1 1 1 1 1 2 2 2 2 2 2 2 2 2 2
$names
[1] "男" "女"サマリの詳細
boxplot()関数が出力するのはテューキーの箱ひげ図で、出力されるサマリは下表の通りです。
| 返り値 | 説明(返り値の内容) |
|---|---|
stats |
下ひげ極値1、四分位数2、上ひげ極値 |
n |
(グループ毎の)データ数 |
conf |
信頼区間3(\(Q_2 \pm 1.58 \times \frac{IQR}{\sqrt n}\)) |
out |
外れ値4(インデックスではなくデータの値) |
group |
外れ値が属するグループ |
names |
グループ名(層別水準名) |
1 下/上ひげ極値とは閉区間\(\big[ Q_1 - 1.5 \times IQR, Q_3 + 1.5 \times IQR \big]\)の中で最も小さい/大きいデータの値です。閉区間の下限/上限値ではありません。
2 四分位数はテューキーの四分位数(fivenum()関数で求めています)になります。
3 95\(\%\)信頼区間です。係数はソースでベタ打ちになっているため引数を用いて信頼区間を変更することはできません。
4 外れ値は閉区間\(\big[ Q_1 - 1.5 \times IQR, Q_3 + 1.5 \times IQR \big]\)の外側にある値です。
閉区間の外側を外れ値とする理由は下図(正規分布における箱ひげ図)を見れば理解できると思います。
箱ひげ図を出力せずにサマリだけを出力したい場合はplot = FALSEを引数に指定します。
返り値だけを出力する
boxplot(ストレス反応得点 ~ 性別, data = PatientStress, plot = FALSE)$stats
[,1] [,2]
[1,] 0.0 0
[2,] 9.0 14
[3,] 19.0 31
[4,] 38.5 55
[5,] 81.0 114
$n
[1] 119 218
$conf
[,1] [,2]
[1,] 14.72727 26.61254
[2,] 23.27273 35.38746
$out
[1] 97 113 86 87 107 113 156 131 139 126 140 124 141 148 126 142
$group
[1] 1 1 1 1 1 1 2 2 2 2 2 2 2 2 2 2
$names
[1] "男" "女"返り値はリスト型変数なので$演算子を用いた参照で特定の値だけを出力することも可能です。
特定の返り値だけを出力する
boxplot(ストレス反応得点 ~ 性別, data = PatientStress, plot = FALSE)$stats [,1] [,2]
[1,] 0.0 0
[2,] 9.0 14
[3,] 19.0 31
[4,] 38.5 55
[5,] 81.0 114下図の青点線が男性の閉区間上限値、赤点線が女性の閉区間上限値、青緑の四角が各データになります。上ひげ極値は閉区間内にあるデータの最大値を取りますので必ずしも閉区間上限値と一致しないことが図から分かります。なお、閉区間下限値はマイナスになることから出力を省略しますが、下ひげ極値は男女ともに閉区間内にあるデータの最小値であるゼロになっていることが分かります。

独立サンプルt検定
R Commanderで独立サンプルt検定(独立標本t検定)を行う場合は、データセットがTidy data(整然データ)5という概念を満たしている必要があります。
以下の条件を満たす表型のデータをTidy dataといい、構造と意味が合致しているのが特徴です。
1. 個々の変数が一つの列をなす
2. 個々の観測が一つの行をなす
3. 個々の値が一つのセルをなす
4. 個々の観測の構成単位の累計が一つの表をなす
なので、神ExcelはTidy dataにはなりませんのでRでそのまま扱うことはできません。
Tidy dataの具体例としてはRに標準で組み込まれているsleepデータセットが挙げられます。sleepデータセットは二種類の睡眠薬の効果 (睡眠時間の増減) に関するデータです。
sleepデータセットを読み込む
R Commanderでsleepデータセットを読み込む手順は下記の通りです。
- [データ]-[パッケージ内のデータ]-[アタッチされたパッケージからデータセットを読み込む…]を実行する
- 開かれたダイアログの[データセット名を入力:]欄に
sleepと入力し - [OK]ボタンをクリックする
- [データセット:]欄が
sleepになっていることを確認する
独立サンプルt検定の実行
R Commanderで読み込んだsleepデータセットに対する独立サンプルt検定を実行する手順は下記の通りです。この手順ではオプション指定を省略していますので、必要に応じてオプションを指定してください。
- [統計量]-[平均]-[独立サンプルt検定…]を実行する
- 開かれたダイアログのグループが
group、目的変数がextraになっていることを確認し - ダイアログの[OK]ボタンをクリックする
- 独立サンプルt検定が実行される
統計量 - 平均 - 独立サンプルt検定
t.test(extra ~ group, alternative = "two.sided",
conf.level = .95, var.equal = FALSE, data = sleep)
Welch Two Sample t-test
data: extra by group
t = -1.8608, df = 17.776, p-value = 0.07939
alternative hypothesis: true difference in means between group 1 and group 2 is not equal to 0
95 percent confidence interval:
-3.3654832 0.2054832
sample estimates:
mean in group 1 mean in group 2
0.75 2.33 対応のあるt検定
R Commanderで対応のあるt検定(関連のあるt検定、従属なt検定)を行うには、独立サンプルt検定で使ったデータセットとは異なり下記のような形式(ワイド形式)のデータセットが必要です。
R Commanderでこのような形式に変換する手順は下記の通りです。
- アクティブデータセットを
sleepデータセットにする - [データ]-[アクティブデータセット]ー[データセットのロングからワイド形式への変換]を実行する
- 開かれたダイアログで
- 被験者ID変数に
IDを指定 - 被験者内要因に
groupを指定 - 出来事に応じて変化する変数に
extraを指定 - 無視する変数は無指定
- 被験者ID変数に
- ダイアログの[OK]ボタンをクリックする
-
sleepデータセットがsleepWideデータセットに変換される
データ - アクティブデータセット - データセットのロングからワイド形式への変換
sleepWide <- RcmdrMisc::reshapeL2W(sleep,
within="group",
id="ID",
varying="extra")R Commanderで上記の逆変換(ワイド->ロング)を変換する手順は下記の通りです。
- アクティブデータセットを
sleepWideデータセットにする - [データ]-[アクティブデータセット]ー[データセットのワイドからロング形式への変換]を実行する
- 開かれたダイアログで
- [反復測定1因子]タブを選択する
-
Level-1でextra.1を選択する -
Level-2でextra.2を選択する-
Level-1ならびにLevel-2が元のgroupの値になります
-
- [Name for the within-subjects factor:]には任意の変数名を設定する
- これが元の
groupに相当します
- これが元の
- ダイアログの[OK]ボタンをクリックする
-
sleepWideデータセットがsleepWideLongデータセットに変換される
データセット名などは[オプション]タブで指定してください。
データ - アクティブデータセット - データセットのワイドからロング形式への変換
sleepWideLong <- RcmdrMisc::reshapeW2L(sleepWide, within="trials",
levels=list(trials=c("Level-1", "Level-2")),
varying=list(response=c("extra.1","extra.2")),
id="id")対応あるt検定の実行
ワイド形式のデータセットを準備し(ここでは上記のNoteの手順で作成したsleepWideデータセットを利用します)。対応のあるt検定を下記の手順で実行します。この手順ではオプション指定を省略していますので、必要に応じてオプションを指定してください。
- 変換された
sleepWideデータセットをアクティブデータにする - [統計量]-[平均]-[対応のあるt検定…]を実行する
- 開かれだダイアログで
- 第1の変数に
extra.1を指定 - 第2の変数に
extra.2を指定
- 第1の変数に
- ダイアログの[OK]ボタンをクリックする
- 対応のあるt検定が実行される
統計量 - 平均 - 対応のあるt検定
Paired t-test
data: extra.1 and extra.2
t = -4.0621, df = 9, p-value = 0.002833
alternative hypothesis: true mean difference is not equal to 0
95 percent confidence interval:
-2.4598858 -0.7001142
sample estimates:
mean difference
-1.58 Rにおけるt検定
R Commanderにおけるt検定は実行コードを見ればわかるようにすべてのt検定をt.test()というRの標準関数で行っています。t.test()関数は引数の指定で様々なt検定に対応しているからです。
t.test()関数の引数
## Default S3 method:
t.test(x, y = NULL, # 標本の指定
alternative = c("two.sided", "less", "greater"),
# 対立仮設の指定
mu = 0, # 平均値の(差の)指定
paired = FALSE, # 独立か対応かの指定
var.equal = FALSE, # 等分散か否かの指定
conf.level = 0.95, ...)
## S3 method for class 'formula'
t.test(formula, # 標本の指定
data, # データセット名の指定
subset, # サブセットの指定
na.action, # NAへの対処方法
alternative = c("two.sided", "less", "greater"),
# 対立仮設の指定
mu = 0, # 平均値の(差の)指定
paired = FALSE, # 独立か対応かの指定
var.equal = FALSE, # 等分散か否かの指定
conf.level = 0.95, ...)指定方法には二通りありますが下側の’formula’形式を使うとTidy data(整然データ)を変形させずに引数の指定だけで四通りのt検定に対応できますので覚えておいて損はないかと思います。
| t検定の種類 | formula | paired | var.equal |
|---|---|---|---|
| 1標本のt検定 | value ~ 1 |
FALSE |
FALSE |
| スチューデントのt検定6 | value ~ group |
FALSE |
TRUE |
| ウェルチのt検定7 | value ~ group |
FALSE |
FALSE |
| 対応のあるt検定 | value ~ group |
TRUE |
FALSE |
6 スチューデントのt検定とは二標本間でデータの母分散が等しいと仮定できるときに用いる検定手法です
7 ウェルチのt検定とは二標本間でデータの母分散が等しいとは限らないときに用いる検定手法です
対立仮設(alternativeオプション)は"two.sided"、"less"、"greater"のどれかを指定します。デフォルトは"two.sided"(両側検定)です。
1標本のt検定の場合
1標本のt検定の場合は標本の平均値(\(\mu\))と比較値(\(\mu_0\))に対する検定になります。
| 対立仮設 | 帰無仮設 | 指定 |
|---|---|---|
| \(\mu \neq \mu_0\) | \(\mu = \mu_0\) | "two.sided" |
| \(\mu \lt \mu_0\) | \(\mu \ge \mu_0\) | "less" |
| \(\mu \gt \mu_0\) | \(\mu \le \mu_0\) | "greater" |
独立サンプルt検定の場合
独立サンプルt検定の場合は各標本の平均値(\(\mu_x\)と\(\mu_y\))に対する検定になります。
| 対立仮設 | 帰無仮設 | 指定 |
|---|---|---|
| \(\mu_x \neq \mu_y\) | \(\mu_x = \mu_y\) | "two.sided" |
| \(\mu_x \lt \mu_y\) | \(\mu_x \ge \mu_y\) | "less" |
| \(\mu_x \gt \mu_y\) | \(\mu_x \le \mu_y\) | "greater" |
対応のあるt検定の場合
対応のあるt検定の場合は二標本の平均の差(\(\delta = \mu_x - \mu_y\))に対する検定になります。
| 対立仮設 | 帰無仮設 | 指定 |
|---|---|---|
| \(\delta \neq 0\) | \(\delta = 0\) | "two.sided" |
| \(\delta \lt 0\) | \(\delta \ge 0\) | "less" |
| \(\delta \gt 0\) | \(\delta \le 0\) | "greater" |
以上